PROGETTO: dalla TAC al modello 3D della gamba
Scopo del progetto: ricostruire in 3D i muscoli della gamba.
Materiale a disposizione: TAC, fettine di 512 x 512 con pixel di circa 1mm x 1mm (volume 60 x 60 cm circa) di tutta la gamba dal bacino al piede.
Obbiettivo: separare in modo automatico all'interno di ciascuna fettina: pelle, grasso, muscolo, osso
(all'interno dell'osso si trova il midollo). A partire dalle sezioni di muscolo sara' possibile ricostruire un modello 3D della muscolatura.
Note: attualmente esiste un sistema che lavora su immagini 2D. Dal piede fino alla parte superiore della coscia la segmentazione 2D viene gia' fatta abbastanza bene utilizzando tecniche di fuzzy clustering (fuzzy C-mean) sulle singole fettine. I problemi si hanno quando si arriva al livello di parte superiore della coscia. La segmentazione separa attualmente osso (+ midollo), muscolo e grasso (+ pelle). A livello di coscia superiore sarebbe opportuno inserire una nuova classe. Come primo passo sarebbe sufficiente implementare un clustering che tenga conto del risultato della segmentazione della fettina successiva. Si puo' pensare ad un tracking della regione del muscolo, ad una inizializzazione della classe muscolo, all'utilizzo di mutua informazione o ad altre tecniche adatte.
Collaborazioni: Istituto di Bioimmagini Molecolari del CNR di Segrate (Milano), referente Ing. Giovanna Rizzo.
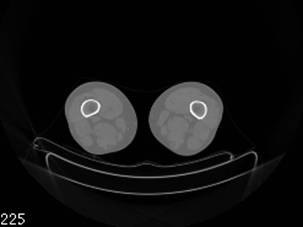
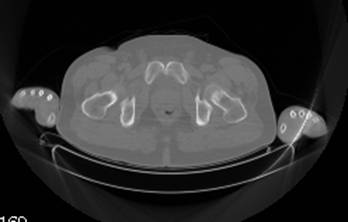
Le immagini riportate sotto (a livelli di grigio) rappresentano due fette TAC ad altezza diversa: appena sotto il ginocchio(a), ad altezza di bacino (b). La facilità di identificare le regioni è evidente. In particolare, le regioni indicate con il bianco sono le regioni ossee, le regioni in grigio chiaro sono i muscoli, in grigio più scuro il grasso. La struttura alla base dell’immagine è il cuscino su cui è appoggiato il soggetto durante l’analisi TAC. Nell’immagine (b) la difficoltà risiede nel tracciare il confine della regione occupata dai singoli muscoli.